Un nuevo modelo de inteligencia artificial puede mejorar el proceso de descubrimiento de fármacos y vacunas al predecir con qué eficiencia secuencias específicas de ARNm producirán proteínas, tanto en general como en diversos tipos de células.
por la Universidad de Texas en Austin
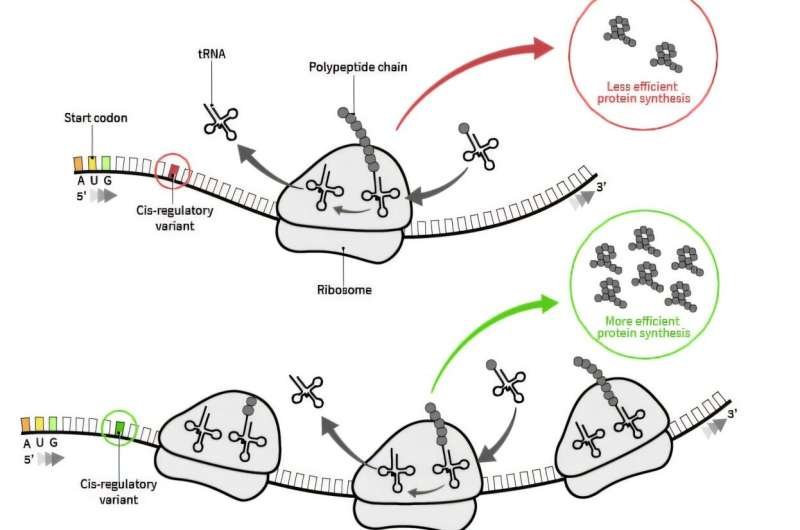
El nuevo avance, desarrollado a través de una asociación académico-industrial entre la Universidad de Texas en Austin y Sanofi, ayuda a predecir cuánta proteína producirán las células, lo que puede minimizar la necesidad de experimentación de prueba y error, acelerando la próxima generación de terapias de ARNm.
El ARN mensajero (ARNm) contiene instrucciones sobre qué proteínas producir y cómo hacerlo, lo que permite que nuestro cuerpo crezca y lleve a cabo las funciones cotidianas de la vida. Entre las áreas más prometedoras de la salud y la medicina, la capacidad de desarrollar nuevas vacunas y fármacos de ARNm —capaces de combatir virus, cánceres y trastornos genéticos— implica el proceso, a menudo complejo, de inducir a las células del cuerpo de un paciente a producir suficiente proteína a partir del ARNm terapéutico para combatir eficazmente las enfermedades.
El nuevo modelo, llamado RiboNN, se propone guiar el diseño de nuevas terapias basadas en ARNm, al determinar qué producirá la mayor cantidad de proteína o qué partes específicas del cuerpo, como el corazón o el hígado, serán más efectivas. El equipo describió su modelo hoy en uno de dos artículos relacionados publicados en la revista Nature Biotechnology .
«Cuando comenzamos este proyecto hace más de seis años, no había ninguna aplicación obvia», dijo Can Cenik, profesor asociado de biociencias moleculares en UT Austin, quien codirigió el trabajo con Vikram Agarwal, jefe de ciencia de datos de diseño de plataformas de ARNm en el Centro de Excelencia de ARNm de Sanofi.
Nos intrigaba saber si las células coordinan los ARNm que producen y con qué eficiencia se traducen en proteínas. Ese es el valor de la investigación impulsada por la curiosidad. Sienta las bases para avances como RiboNN, que solo serán posibles mucho más tarde.
En pruebas que abarcaron más de 140 tipos de células humanas y de ratón, RiboNN mostró una precisión de aproximadamente el doble en la predicción de la eficiencia de la traducción que los enfoques anteriores. Este avance podría brindar a los investigadores la capacidad de realizar predicciones en células que podrían ayudar a agilizar los tratamientos contra el cáncer y las enfermedades infecciosas y hereditarias.
Puedes imaginar la forma en que las células de tu cuerpo producen proteínas como si un equipo de chefs horneara pasteles. Para preparar un lote de proteínas, los chefs de una de tus células (ribosomas) buscan la receta en tu recetario de proteínas (también conocido como ADN), la copian en fichas llamadas ARN mensajeros (ARNm) y luego combinan los ingredientes (aminoácidos) según la receta para hornear los pasteles (proteínas).
Una vacuna o terapia de ARNm induce a estos chefs celulares a producir proteínas. En el caso de una vacuna, podrían producir una proteína presente en la superficie de un virus patógeno o de células cancerosas, lo que básicamente alerta al sistema inmunitario para que produzca anticuerpos contra el virus o el cáncer.
En el caso de un trastorno causado por una mutación genética, podrían producir una proteína que el cuerpo no puede producir adecuadamente por sí solo, revirtiendo el trastorno.
Antes de desarrollar su nuevo modelo predictivo, Cenik y el equipo de UT primero seleccionaron un conjunto de datos disponibles públicamente de más de 10.000 experimentos que medían la eficiencia con la que diferentes ARNm se traducen en proteínas en distintos tipos de células humanas y de ratón.
Una vez creado este conjunto de datos de entrenamiento, los expertos en inteligencia artificial y aprendizaje automático de UT y Sanofi se unieron para desarrollar RiboNN.
Uno de los objetivos de la herramienta predictiva es algún día crear terapias dirigidas a un tipo de célula en particular, dijo Cenik, quien también es miembro afiliado del cuerpo docente del Instituto Oden de Ingeniería Computacional y Ciencias de la UT y académico del CPRIT, que recibe apoyo de investigación del Instituto de Prevención e Investigación del Cáncer de Texas.
«Quizás se necesite una terapia de nueva generación que se desarrolle en el hígado, el pulmón o las células inmunitarias», dijo. «Esto abre la oportunidad de modificar la secuencia del ARNm para aumentar la producción de esa proteína en ese tipo celular».
En un artículo complementario, también publicado en Nature Biotechnology , el equipo demostró que los ARNm con funciones biológicas relacionadas se traducen en proteínas en niveles similares en diferentes tipos celulares. Los científicos saben desde hace tiempo que el proceso de transcripción de genes con funciones relacionadas en ARNm está coordinado, pero no se había demostrado previamente que la traducción de ARNm a proteínas también lo esté.
Los estudiantes de pregrado investigadores de la UT verificaron manualmente la precisión de los datos disponibles y completaron la información faltante para crear RiboBase, el conjunto de datos necesario para entrenar el modelo de IA.
Los equipos que colaboraron para desarrollar RiboNN incluyeron a Logan Persyn, estudiante de posgrado en informática de la UT, y a Dinghai Zheng y Jun Wang, de Sanofi. La oficina «Descubrimiento al Impacto» de la UT facilitó la colaboración entre la UT y Sanofi mediante el desarrollo de un acuerdo de investigación.
Más información: Zheng, D. et al. Predicción de la eficiencia de la traducción del ARN mensajero en células de mamíferos, Nature Biotechnology (2025). DOI: 10.1038/s41587-025-02712-x
Liu, Y. et al. La covariación de la eficiencia de la traducción identifica patrones de coordinación conservados entre tipos celulares. Nature Biotechnology (2025). doi.org/10.1038/s41587-025-02718-5
Nota editorial:
Este artículo ha sido elaborado con fines divulgativos a partir de información pública y fuentes especializadas, adaptado al enfoque editorial del medio para facilitar su comprensión y contextualización.
