A medida que aumentan las temperaturas globales, los científicos del Laboratorio Cold Spring Harbor (CSHL) trabajan para desarrollar cultivos más fuertes y resilientes. Sin embargo, este proceso es un desafío.
por Margaret Osborne, Laboratorio Cold Spring Harbor
Las plantas suelen tener varios genes relacionados que controlan características deseables, como el tamaño o la resistencia a la sequía. Encontrar genes con funciones superpuestas, o «genes redundantes», es una búsqueda casi imposible.
«La mayoría de las veces, existen importantes limitaciones en el proceso de mejora de los cultivos», afirmó Iacopo Gentile, investigador postdoctoral del laboratorio Zachary Lippman del CSHL. «Esto se debe a la gran redundancia y complejidad en la evolución y compensación de las familias de genes».
Ahora, Gentile y sus colegas han rastreado una importante familia de genes en plantas con flores para ver cómo ha cambiado a lo largo de 140 millones de años de evolución. Con estos datos, entrenaron modelos para identificar patrones de redundancia y predecir qué genes editar para modificar rasgos específicos. Los hallazgos se publican en la revista Molecular Biology and Evolution .
«Se trata de comprender qué sucede tras la duplicación genética», explicó Gentile. «Un gen se duplica. Luego dos. ¿Qué sucede después? La teoría indica que divergirán. La gran incógnita en este campo es cómo».
Para responder a esta pregunta, el equipo se centró en el CLE , una familia de genes implicada en la señalización celular y el desarrollo vegetal. Los péptidos CLE son prevalentes en todas las especies vegetales. Sin embargo, se desconoce mucho sobre sus funciones específicas. Su estudio ha sido difícil debido a su corta longitud, rápida evolución y redundancia.
Utilizando nuevos avances en IA , el equipo identificó miles de genes CLE previamente desconocidos en 1000 especies. Introdujeron estos datos en modelos informáticos, que identificaron genes que podrían ser redundantes. Los genes redundantes probablemente comparten similitudes en uno o dos aspectos: los péptidos que producen o los promotores genéticos, las áreas del ADN que controlan la expresión.
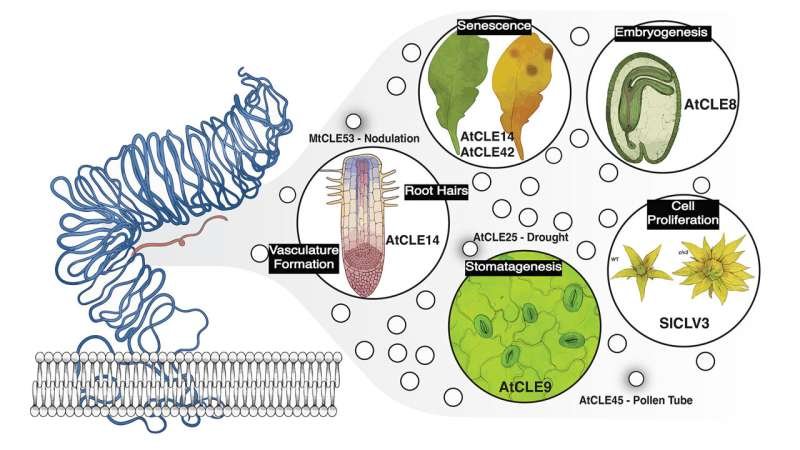
Para confirmar las predicciones de los modelos , el laboratorio de Lippman eliminó los genes marcados en tomates mediante CRISPR. Como se sospechaba, eliminar solo una redundancia no tuvo ningún efecto. Sin embargo, eliminarlos todos produjo cambios visibles en las plantas.
«Es la primera vez en tomates que se logra una focalización tan amplia de tantos genes al mismo tiempo», dijo Gentile. «Nos enfocamos en 10».
Cabe destacar que el equipo descubrió que la mayoría de los genes redundantes tenían promotores similares, incluso si las secuencias peptídicas diferían. El modelo no solo identificó posibles redundancias, sino que también predijo si mutaciones específicas de CLE tendrían efectos positivos, negativos o neutrales en las plantas.
Gentile afirmó que el método que desarrollaron podría «escalarse fácilmente a todas las familias de genes», no solo a la CLE. Como resultado, los fitomejoradores ahora cuentan con una «hoja de ruta» para predecir cómo podrían aprovecharse de los genes ocultos.
Más información: Iacopo Gentile et al., El análisis panangiospermano de la familia de genes del péptido de señalización CLE revela rutas, patrones y predicciones de diversificación de parálogos, Biología Molecular y Evolución (2025). DOI: 10.1093/molbev/msaf294
