La secuencia del genoma de un reptil inusual llamado tuatara arroja luz sobre la evolución de la especie y las estrategias de conservación. El trabajo es un modelo de las mejores prácticas actuales para colaborar con las comunidades indígenas.

Rebecca N. Johnson
Una orden de reptiles una vez rica en especies llamada Rhynchocephalia vivió en todo el mundo durante la época de los dinosaurios 1 , 2 . Solo una de estas especies sobrevive hoy: la tuatara (Fig. 1).
Solo se encuentran en Nueva Zelanda, los tuatara son un taonga (‘tesoro especial’) para los maoríes. Los reptiles tienen un conjunto de rasgos intrigantes, incluida la longevidad y una combinación inusual de características morfológicas similares a las de aves y reptiles 3 , que han llevado a la incertidumbre sobre su lugar en el árbol evolutivo.
Writing in Nature , Gemmell et al. 4 informan la primera secuencia de genoma completo para la tuatara ( Sphenodon punctatus) El estudio de los investigadores proporciona información sobre la biología y la evolución de este animal extraordinario.

El trabajo es una colaboración entre genomicistas y Ngātiwai, los iwi maoríes (personas) que tienen la custodia de las poblaciones de tuatara utilizadas en este estudio. Incluso con los avances en la tecnología de secuenciación del genoma en los últimos años, no es posible producir una secuencia del genoma de alta calidad sin acceso a un buen material genético. Los investigadores obtuvieron esto solo a través de la colaboración. Ngātiwai participó en todos los procesos de toma de decisiones para este estudio, y se enumeran como los últimos autores del artículo. Gemmell y col.también proporcionan un modelo de acuerdo que otros investigadores pueden seguir si desean consultar con los guardianes tradicionales de otros organismos. Como tal, el estudio establece un nuevo estándar para la colaboración con los guardianes indígenas en genómica y otros esfuerzos científicos.
El genoma producido por Gemmell y sus colaboradores es uno de los genomas de vertebrados más grandes publicados hasta ahora. Con más de 5 gigabases, es aproximadamente un 50% más grande que el genoma humano. Para complementar el genoma, los autores generaron perfiles de expresión génica para sangre de tuatara y embriones. También realizaron un análisis preliminar de secciones activas e inactivas del genoma, y un análisis en profundidad de regiones repetidas. El genoma representa un recurso valioso para futuras investigaciones sobre una variedad de temas, desde la evolución de la puesta de huevos hasta por qué la Rhynchocephalia, que alguna vez fue rica en especies, tiene un solo sobreviviente.
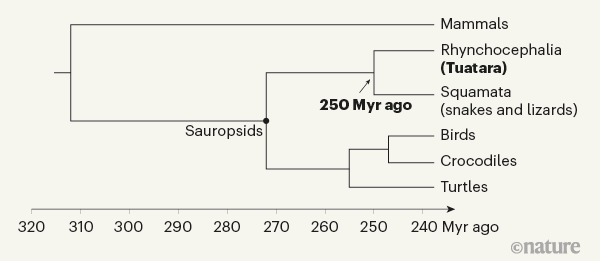
Una razón para secuenciar genomas es reconstruir el árbol evolutivo de la vida; esto permite una comprensión más profunda de cómo evolucionó la vida, y este conocimiento se puede utilizar para abordar desafíos como la pérdida de biodiversidad y el cambio climático. Gemmell y col. utilizó métodos de genómica comparativa para hacer precisamente eso. Generaron un árbol filogenético para el Sauropsida (un clado que incluye a todos los reptiles modernos, junto con las aves) comparando las secuencias del genoma de 27 vertebrados, incluida la tuatara (Fig. 2). El árbol de investigadores confirma una sugerencia previa 5 de que la Rhynchocephalia divergió de sus parientes más cercanos 5 , 6, los Squamata (lagartos y serpientes), hace unos 250 millones de años, durante el período Triásico Temprano. La confirmación de una divergencia tan temprana es importante para comprender el origen y la evolución de la Lepidosauria, que comprende tanto la Rhynchocephalia como la Squamata.
¿Podría la tuatara ser un fósil viviente? El término, que se refiere a una especie que ha evolucionado extremadamente lento y aún conserva las características de sus ancestros antiguos, ha caído en desgracia con los paleontólogos y biólogos evolutivos. Esto se debe, en parte, al uso indebido del término, que puede surgir cuando falta evidencia fósil que hubiera reflejado cambios físicos en una especie, o cuando los investigadores asumen erróneamente que un único sobreviviente de un linaje dado debe haber permanecido sin cambios durante la evolución. hora. Los tuatara se parecen mucho a sus antepasados de la era mesozoica temprana 7 , hace entre 240 y 230 millones de años. Sin embargo, no existe un registro fósil continuo 6, lo que dificulta definir qué rasgos podría compartir el tuátara con sus ancestros ahora extintos.
La reconstrucción filogenética de Gemmell y sus colegas indica que el tuátara tiene la tasa de evolución más baja de cualquier lepidosaurio descrito hasta ahora. Estos datos podrían sugerir que la tuatara es de hecho un fósil viviente. Además de su largo tiempo de generación y su baja temperatura corporal, la lenta evolución del tuatara podría hacerlo particularmente vulnerable a un clima más cálido.
Luego, los autores analizaron el genoma del tuatara con más detalle. En promedio, más del 50% del genoma de un vertebrado está compuesto por secuencias de ADN repetitivas (elementos repetidos) 8 , 9 . De acuerdo con esta cifra, el 64% del genoma de tuatara son elementos repetidos. Sin embargo, los tipos de elementos repetidos fueron una combinación de similares a mamíferos y reptiles. Este es un hallazgo clave, porque el ancestro común más reciente de los sauropsidos fue imputado como un reptil sobre la base de las características genómicas encontradas en las aves y lagartos, algunos de los cuales tienen genomas muy bien caracterizados 10 . Al revelar características inesperadas, similares a las de los mamíferos, el genoma de la tuatara proporciona nuevas perspectivas evolutivas.
Los investigadores también encontraron que el genoma de tuatara tiene una gama más amplia de secuencias de ADN llamadas transposones (secuencias que pueden moverse de una ubicación genómica a otra) que cualquier otro reptil, pájaro o mamífero secuenciado hasta ahora. Muchos de estos parecen haber estado activos recientemente (probablemente en los últimos millones de años), lo que sugiere que todavía tienen o han tenido un papel recientemente en la configuración del genoma. Los autores sugieren que el gran genoma del tuatara podría explicarse por el hecho de que casi un tercio de él consiste en duplicaciones de secuencias de ADN entre 1 y 400 kilobases de largo.
Gemmel y col. luego comparó los genes de tuatara asociados con la vista, el olfato, la inmunidad, la termorregulación y la longevidad con los genes equivalentes en otras especies. A pesar de ser nocturno, la tuatara es un depredador altamente visual, y los autores encontraron evidencia de que ha retenido genes asociados con la visión que recuerdan a un ancestro que estuvo activo durante el día. La especie parece haber conservado una visión robusta del color, incluso con niveles de luz bajos, lo que sugiere que podría haber un beneficio adaptativo al tener este rasgo.
Además, la tuatara parece tener un repertorio de varios cientos de receptores de olores, similar al número de aves, pero más bajo que el de los cocodrilos o las tortugas. Se requieren más investigaciones para investigar la función de estos receptores y determinar las implicaciones de este repertorio de receptores reducido para la alimentación y la caza de tuataras. Por ejemplo, quizás los tuatara dependen de su visión para cazar (como las aves), en lugar de depender de los olores y otros sentidos (como las serpientes).
Finalmente, hay un debate en curso sobre si realmente hay dos subespecies de tuatara: información crucial para las estrategias de conservación. Debido a que los animales están protegidos, los autores evaluaron la diversidad genética entre la población utilizando muestras recolectadas durante muchas décadas. Este análisis confirma que solo hay una especie de tuátara, a pesar de que una población (en la isla North Brother en el estrecho de Cook) es genéticamente distinta de las demás. La falta de muestras actuales no es deseable para diseñar enfoques de conservación basados en la genética, pero, dada la longevidad del tuátara, es probable que las recomendaciones que surjan del estudio sigan siendo válidas.
Aunque la secuenciación del genoma completo ha beneficiado la salud humana y ha mejorado nuestra comprensión de la evolución humana, la secuenciación de genomas de otros organismos puede tener muchos beneficios, especialmente para aquellos organismos que enfrentan la pérdida de biodiversidad causada por los humanos. Sin embargo, para muchas de estas especies, las muestras no están fácilmente disponibles. El trabajo de Gemmell y sus colegas nos recuerda que la recolección de muestras y la consulta con los pueblos indígenas pueden ir de la mano para mejorar los resultados de la conservación biológica y cultural.doi: 10.1038 / d41586-020-02063-4
Referencias
1)Evans, SE Biol. Rev. Camb. Phil Soc. 78 , 513–551 (2003).
2)Hsiou, AS y col. Sci. Rep. 9 , 11821 (2019).
3)Gunther, A. Phil. Trans. R. Soc. Lond. 157 , 595-629 (1867).
4)Gemmell, NJ y col. Naturaleza https://doi.org/10.1038/s41586-020-2561-9 (2020).
5)Jones, MEH y col. BMC Evol. Biol. 13 , 208 (2013).
6)Jones, MEH y Cree, A. Curr. Biol. 22 , R986 – R987 (2012).
7)Herrera-Flores, JA, Stubbs, TL y Benton, MJ Palaeontology 60 , 319–328 (2017).
8.Pasquesi, GIM y col. Nature Commun. 9 , 2774 (2018).
9)Sotero-Caio, CG, Platt, RN II, Suh, A. y Ray, DA Genome Biol. Evol. 9 , 161-177 (2017).
10.Zhang, G. y col. Science 346 , 1311-1320 (2014).
