La lechuga (L. sativa L., 2n = 2x = 18) es una verdura de hoja importante desde el punto de vista agrícola que se cultiva predominantemente en climas templados y se produce en grandes cantidades en Corea.
por la Universidad Agrícola de NanJing
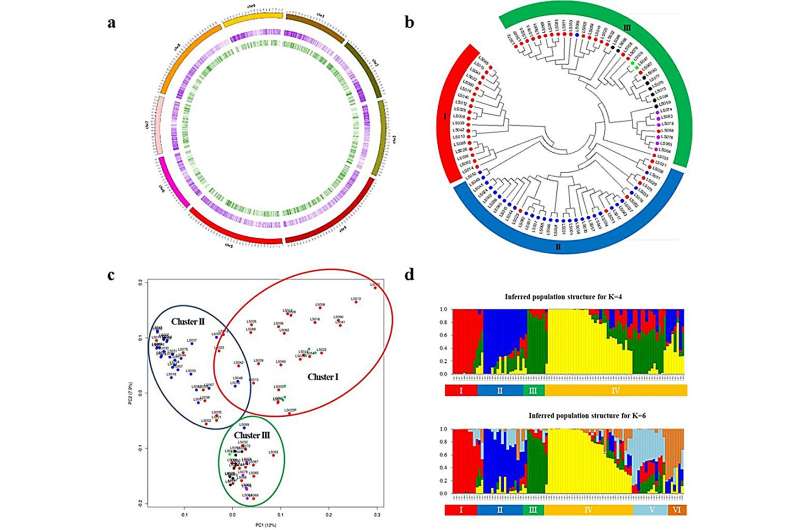
La decodificación completa de su genoma y el análisis de alrededor de 45.000 genes han permitido avanzar en el desarrollo de cultivares. Sin embargo, distinguir cultivares de lechuga estrechamente relacionados sigue siendo un desafío, particularmente debido a su diversidad morfológica y perfiles genéticos similares.
Los métodos tradicionales, como la prueba de distinción, uniformidad y estabilidad (DHE) basada en características morfológicas, requieren mucha mano de obra y están sujetos a condiciones ambientales. Por lo tanto, la aplicación y mejora de marcadores moleculares como SSR y SNP para el análisis genético es necesaria para una identificación de cultivares de lechuga y estudios genéticos más precisos y eficientes .
En mayo de 2022, Horticulture Research publicó un artículo de investigación titulado » Conjuntos básicos de marcadores SNP y ensayos Fluidigm de todo el genoma para una identificación genotípica rápida y eficaz de cultivares coreanos de lechuga (Lactuca sativa L.) «.
En este estudio, los investigadores desarrollaron y validaron conjuntos básicos de marcadores de polimorfismo de un solo nucleótido (SNP) de todo el genoma para la identificación de cultivares de lechuga mediante enfoques de genotipado por secuenciación (GBS) y genotipado SNP, seguidos de un análisis de alto rendimiento en la plataforma Fluidigm. .
GBS identificó 17.877 SNP de alta calidad para 90 cultivares comerciales de lechuga. Utilizando estos SNP, el análisis filogenético clasificó los 90 cultivares en tres grupos distintos, alineándose en gran medida con sus tipos hortícolas. El análisis de componentes principales (PCA) y el análisis de la estructura poblacional utilizando estos SNP arrojaron resultados de agrupamiento similares, reforzando así la estrecha asociación entre los genotipos de SNP y los tipos hortícolas.
Para desarrollar marcadores de SNP eficaces para la identificación de cultivares, se seleccionaron 294 SNP con polimorfismo significativo. El conjunto básico de 192, 96, 48 y 24 marcadores se analizó y validó adicionalmente utilizando la plataforma Fluidigm. El análisis filogenético basado en el conjunto básico de todos los SNP diferenció con éxito los cultivares identificados hasta ahora.
Estos conjuntos básicos de marcadores SNP serán útiles para construir una base de datos de ADN de lechuga para la identificación de cultivares y pruebas de pureza, así como para respaldar el examen DHE (distinción, uniformidad y estabilidad) en el marco del sistema de protección de obtenciones vegetales (PVP) basado en la UPOV. Finalmente, el estudio validó los 24 marcadores centrales con genotipado Fluidigm en mezclas de ADN, demostrando su capacidad para identificar líneas tanto homocigotas como heterocigotas.
En resumen, este estudio desarrolló y validó con éxito conjuntos básicos de marcadores SNP para la identificación de cultivares de lechuga mediante genotipado por secuenciación (GBS). Se espera que este enfoque mejore significativamente la eficiencia y precisión de la identificación de cultivares de lechuga , proporcionando una herramienta valiosa para futuras investigaciones genéticas y aplicaciones agrícolas prácticas.
Más información: Jee-Soo Park et al, Conjuntos básicos de marcadores SNP y ensayos Fluidigm para todo el genoma para una identificación genotípica rápida y eficaz de cultivares coreanos de lechuga (Lactuca sativa L.), Horticulture Research (2022). DOI: 10.1093/hora/uhac119
