La naranja dulce (Citrus sinensis L.), un cultivo frutal híbrido procedente de mandarinas (Citrus reticulata Blanco) y pomelos [Citrus maxima (Burm.) Merr.], exhibe una compleja diversidad genómica debido a una antigua hibridación interespecífica. En la actualidad, el mejor ensamblaje del genoma de la naranja dulce es la naranja dulce doble haploide (HSO).
por la Universidad Agrícola de NanJing
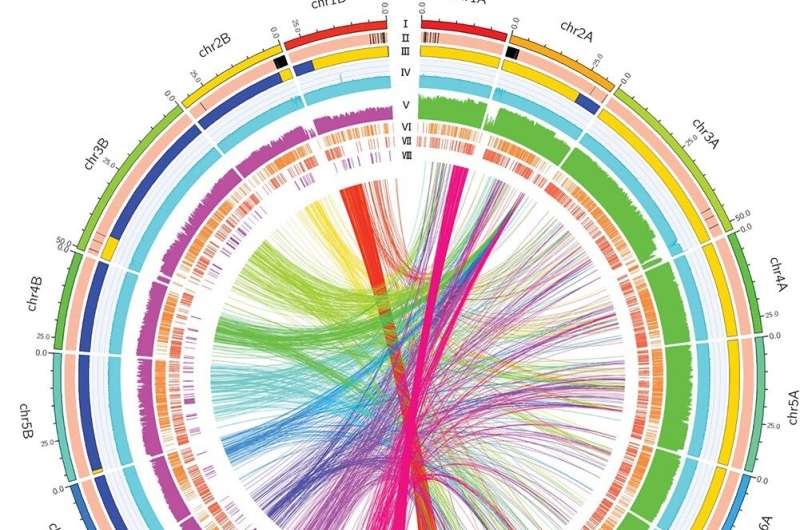
La secuenciación del genoma diploide de la naranja dulce solo produjo ensamblajes de tamaño haploide de mala calidad, que no proporcionaron un nivel de referencia del genoma. La uniformidad de los cultivares de naranja dulce los hace propensos a enfermedades como el Huanglongbing (HLB), que desafía la producción de cítricos a nivel mundial. Por lo tanto, existe una necesidad urgente de ensamblar mejor el genoma a nivel cromosómico de las naranjas dulces y explorar genes para la tolerancia al HLB, lo cual es crucial para la plantación sostenible y el futuro de las naranjas dulces.
En noviembre de 2022, Horticulture Research publicó una perspectiva titulada » Un genoma en fases a nivel de cromosoma que permite estudios a nivel de alelos en naranja dulce: un estudio de caso sobre la tolerancia de los cítricos al Huanglongbing «.
Para ensamblar un nuevo genoma de naranja dulce, los investigadores emplearon lecturas largas y continuas de PacBio para decodificar el genoma DVS y obtuvieron un ensamblaje de 607,6 Mb con una longitud N50 de 15,4 Mb usando CANU. Luego se aplicó un enfoque de ensamblaje por fases, dejando 3,9 Mb de homocigosidad sin fase.
El ensamblaje final conectó cóntigos en 18 pseudocromosomas de 598,6 Mb, que se clasificaron en conjuntos de cromosomas DVS_A y DVS_B, revelando regiones homólogas que se originan predominantemente en mandarina y pomelo, respectivamente. La tasa de error de la asamblea fue significativamente menor que la HSO previamente establecida. La comparación cromosómica de DVS_A y DVS_B con pomelo y mandarinas mostró un 97,1% de regiones de origen de mandarina en DVS_A y un 87,4% de regiones de origen de pomelo en DVS_B.
La similitud de nucleótidos entre DVS_A y DVS_B fue del 96,2%, identificándose numerosas variaciones. La anotación genética de DVS indicó 55.745 genes que codifican proteínas y la mayor integridad de BUSCO entre los genomas de los cítricos. Los patrones de expresión alélica (AEP) se estudiaron utilizando datos de RNA-seq, y DVS sirvió como referencia superior, con tasas de mapeo significativas. Varios factores, incluido el tipo de tejido, la etapa de desarrollo y la enfermedad, alteraron notablemente los AEP, lo que subraya la complejidad de la expresión genética en las naranjas dulces.
Los investigadores utilizaron además la secuenciación del genoma completo para identificar los mecanismos moleculares que contribuyen a la tolerancia al Huanglongbing (HLB) en un mutante de naranja dulce. Los resultados mostraron que, aunque las infecciones del T19 tolerante a la enfermedad eran equivalentes a las de los sensibles T78 y DVS, los árboles T19 tenían un índice de área foliar más alto y un crecimiento más saludable.
Los árboles T19 y un espécimen SF con etiqueta perdida que se confirmó que eran genéticamente idénticos a T19 no mostraron variaciones estructurales somáticas (SV) y compartieron tres SV somáticas. Estas variaciones implicaron translocaciones e inserciones complejas, que afectaron a varios genes y contribuyeron potencialmente a la tolerancia al HLB. Una investigación adicional sobre la expresión alélica mostró una regulación positiva y negativa significativa de los alelos en T19 en comparación con DVS y T78.
Hubo un cambio significativo en la proporción de expresión de alelos sesgados y alelos en DVS, T19 y T78. T19 demostró patrones de regulación positiva únicos en los genes de respuesta al estrés, el metabolismo mitocondrial y otras áreas no observadas en DVS o T78. Esto fue respaldado por perfiles transcriptómicos y la presencia de microARN expresados diferencialmente en T19.
Además, T19 tenía proteínas de choque térmico (HSP) significativamente más reguladas al alza, que son cruciales para la tolerancia al estrés, y genes implicados en la reducción de las especies oxidativas reactivas, que se sabe que alivian los síntomas del HLB. Los hallazgos sugirieron que estas diferencias transcriptómicas, probablemente resultantes de mutaciones somáticas, subyacen a la tolerancia superior al HLB en T19, principalmente a través de respuestas mejoradas al estrés y la protección de la homeostasis de las proteínas del floema.
En conclusión, los investigadores han ensamblado un genoma diploide en fases de alta precisión de la naranja dulce Valencia, lo que ha permitido realizar estudios a nivel de alelos sin precedentes en la genética de los cítricos. Este ensamblaje genómico no sólo aclara la compleja genética de las naranjas dulces, sino que también promete guiar futuros programas de ingeniería genética y mejoramiento de variedades de cítricos mejoradas.
Más información: Bo Wu et al, Un genoma en fases a nivel de cromosoma que permite estudios a nivel de alelos en naranja dulce: un estudio de caso sobre la tolerancia de los cítricos a Huanglongbing, Horticulture Research (2022). DOI: 10.1093/hora/uhac247
