Como fuerza impulsora importante de la evolución, la poliploidía (duplicación del genoma) es omnipresente en las diferentes etapas evolutivas del árbol de la vida de las plantas con flores.
por Zhang Nannan, Academia China de Ciencias
Sin embargo, las interacciones entre los genomas ancestrales en un núcleo poliploide, que a menudo implican dominancia subgenomática, no se han comprendido bien.
Los bambúes muestran una gran diversidad en especies y morfología, y consisten en un clado herbáceo menor, en su mayoría diploide (~126 especies) y tres clados leñosos poliploides principales (~1576 especies). Como uno de los linajes de plantas más importantes que se ha adaptado a un hábitat forestal, los bambúes leñosos (WB) exhiben rasgos biológicos distintivos, como un crecimiento rápido (hasta 114,5 cm/día) y una floración sincrónica, generalmente monocárpica (~30– 60 años).
Un equipo de investigación dirigido por el profesor Li Dezhu del Instituto de Botánica de Kunming (KIB) de la Academia de Ciencias de China (CAS), en colaboración con científicos de Estados Unidos, ha propuesto un modelo refinado para el origen y la poliploidización del bambú leñoso. . Los resultados de su trabajo se publicaron en línea en Nature Genetics en un artículo titulado «Los conjuntos de genomas de 11 especies de bambú resaltan la diversificación inducida por la dominancia dinámica del subgenoma».
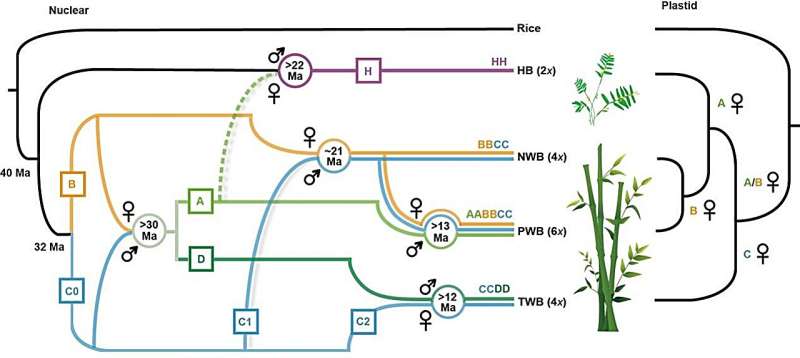
Sus estudios revelaron eventos de hibridación recurrentes entre ancestros diploides de linajes leñosos seguidos de poliploidización, junto con introgresión entre linajes ancestrales leñosos y herbáceos, en las primeras etapas de la evolución del bambú.
El equipo de Li se ha dedicado durante mucho tiempo a estudios sistemáticos y evolutivos del bambú. En su trabajo anterior que informaba borradores de genomas para cuatro especies de bambú, propusieron un origen reticulado para los WB que implica hibridación y poliploidización.
«Para realizar un estudio más completo de los bambúes en diferentes niveles de ploidía y una amplia diversidad filogenética, tomamos muestras de dos bambúes herbáceos y tres especies representativas de cada linaje leñoso», dijo Li.
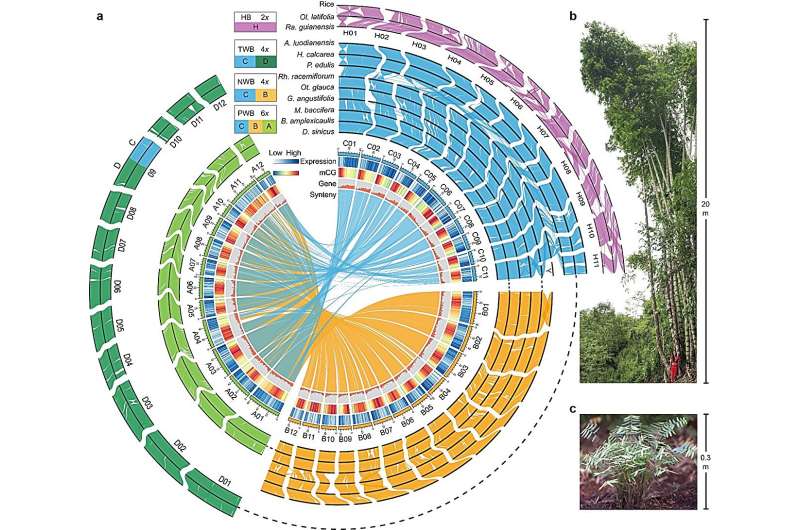
El equipo internacional adquirió con éxito secuencias del genoma a nivel de cromosomas de 11 especies de bambú representativas utilizando tecnología de secuenciación de tercera generación. Recolectaron y secuenciaron datos del transcriptoma de 476 muestras de bambú que cubrían diferentes tejidos y diferentes etapas de crecimiento.
Utilizando un enfoque multiómico, el equipo confirmó la presencia de cuatro subgenomas distintos de WB: A, B, C y D.
Investigaciones adicionales revelaron que los WB exhiben una notable estabilidad del cariotipo, manteniendo el estado ancestral de 12 cromosomas de la familia de las gramíneas a nivel del subgenoma, a pesar de 12 a 20 Ma desde la poliploidización y la posterior diversificación de especies a gran escala.
«Nuestro estudio sugiere un claro dominio de los subgenomas C en dos linajes tetraploides, como se refleja en una serie de características, incluido el tamaño del genoma , el fraccionamiento de genes, los reordenamientos genómicos, los elementos transponibles y la expresión genética», dijo Li.
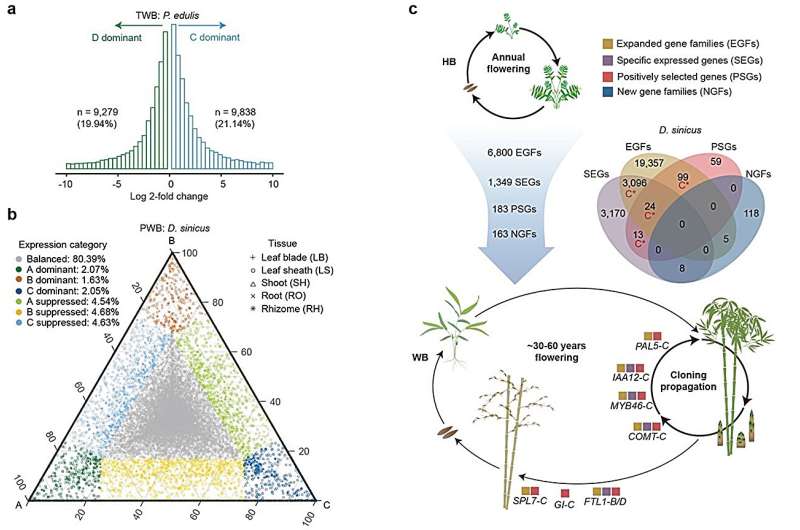
Sin embargo, el patrón de dominancia en el linaje hexaploide es más dinámico, con un cambio gradual en la dominancia del subgenoma C al A.
Un análisis más detallado reveló que el subgenoma C dominante, junto con el subgenoma A en el clado hexaploide, contribuyó más a la evolución de rasgos distintivos en los WB y posiblemente a su radiación adaptativa en un hábitat forestal .
Este trabajo destaca la utilidad de utilizar conjuntos de secuencias del genoma de todo el clado para avanzar en nuestra comprensión de la evolución del subgenoma en plantas poliploides.
Más información: Peng-Fei Ma et al, Los conjuntos de genomas de 11 especies de bambú destacan la diversificación inducida por la dominancia dinámica del subgenoma, Nature Genetics (2024). DOI: 10.1038/s41588-024-01683-0
