A medida que la inseguridad alimentaria mundial alcanzó un nivel peligroso en 2022, los científicos intensificaron sus esfuerzos para perfeccionar las mejores prácticas para proteger los rendimientos de los principales cultivos que son esenciales para combatir este problema.
Y, si bien el arroz constituye una pequeña porción de la cosecha anual de Missouri, junto con el maíz y la soya, son alimentos básicos clave que ayudan a abordar la inseguridad alimentaria no solo en los Estados Unidos, sino en todo el mundo.
En un estudio reciente que examinó cómo funcionan las enfermedades en los cultivos de arroz, los investigadores de la Universidad de Missouri podrían haber encontrado respuestas críticas.
VER: ¿Se puede reconciliar la edición de genes con la agricultura ecológica?
En este estudio, Bing Yang, profesor de biología vegetal en la Facultad de Agricultura, Alimentos y Recursos Naturales de MU y el Centro de Ciencias Vegetales Donald Danforth, utilizó la edición del genoma como una herramienta para identificar patógenos problemáticos presentes en ciertas bacterias que conducen a infecciones prolíficas en el arroz . cultivos. Su investigación ayuda a los científicos a comprender cómo funcionan estos patógenos y, por lo tanto, pueden determinar cómo protegerse contra infecciones generalizadas que destruyen los rendimientos.
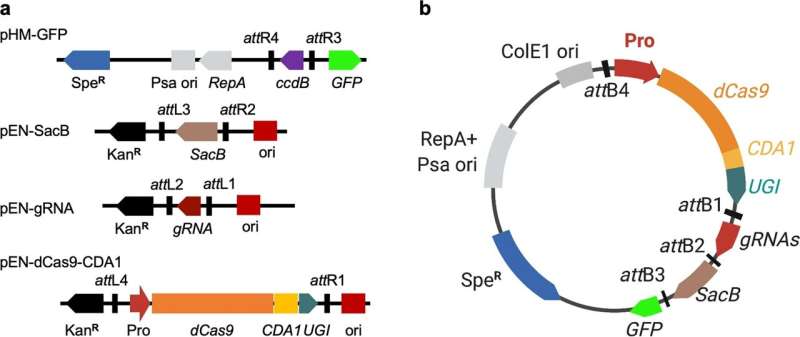
Sistema editor de base de citidina bacteriana fitopatógena. a Un sistema de cuatro módulos para hacer un editor de bases de citidina específico de genes. Los casetes de expresión de ARN guía (ARNg), SacB y el gen quimérico de Cas9 muerto (dCas9), citidina desaminasa (CDA1) e inhibidor de uracilo glicosilasa de ADN (UGI) están flanqueados por motivos de recombinación Gateway (att R y att L). b Mapa esquemático de una sola construcción que expresa Cas9 desaminasa muerta, casete de expresión de ARN guía y GFP. Pro, promotor derivado de Xanthomonas . dCas9, una nucleasa muerta Cas9; CDA1 P. marinus citidina desaminasa, UGI uracilo inhibidor de la ADN glicosilasa, ARN guía de ARNg, SacBel marcador contraseleccionable para el curado del plásmido después de la edición y GFP impulsado por el promotor glT de E. coli . El plásmido contiene el gen resistente a la espectinomicina (SpeR ) , el origen de pSa y RepA, origen de alto número de copias de ColE1. Crédito: Biología de las Comunicaciones (2023). DOI: 10.1038/s42003-023-04451-8
Esta investigación proporciona información sobre la relación huésped-patógeno, lo que permite a los científicos diseñar mejor las plantas genéticamente para sobrevivir a las enfermedades de los cultivos.
«Con base en los avances en la comprensión científica logrados durante este estudio, ahora podemos desarrollar estrategias para diseñar la resistencia del huésped contra la bacteria», dijo Yang. «Así es como podemos apoyar la resistencia de las plantas en general».
Primero, el equipo de investigación descubrió una forma de «eliminar» los genes . Cuando se eliminan genes específicos de la bacteria, los científicos pueden comprender mejor las funciones de esos genes específicos . Luego, los investigadores probaron las propiedades infecciosas, algo que históricamente ha sido un proceso laborioso.
«Esta investigación nos permite comprender mejor qué bacterias tienen cualidades patógenas y cómo esas cualidades corresponden a infecciones en ciertas especies de plantas», dijo Yang. «En última instancia, estos avances en la edición de genes nos ayudan a modificar el genoma de los cultivos, en este caso el arroz, de manera que generen resistencia que los proteja de las enfermedades».
VER: El equipo logra la primera edición de genes de precisión en miscanthus
Utilizando una técnica revolucionaria de edición de genes llamada CRISPR, un método en el que los científicos editan genes cortando el ADN y luego dejándolo reparar de forma natural, Yang y su equipo editaron una muestra de bacterias con el objetivo de determinar exactamente qué genes tenían cualidades patógenas que infectarían las proteínas. en el genoma del cultivo del arroz.
En particular, el método de Yang revoluciona un proceso conocido como recombinación homóloga, que se sabe que es ineficaz y requiere mucho tiempo.
«Mi objetivo de investigación a largo plazo es una mejor comprensión de la biología de las enfermedades y la biología de las plantas y el proceso de uso de tecnología avanzada para diseñar la resistencia a las enfermedades», dijo Yang. «También quiero diseñar el producto para que sea más nutritivo y de una mayor cantidad mientras aumenta el rendimiento de una temporada y reduce la pérdida de rendimiento».
Como estudioso de patología vegetal durante más de 15 años, Yang dijo que las bacterias, incluidas aquellas con cualidades simbióticas (beneficiosas) y patógenas, son esenciales para mantener la vida y la salud de nuestros ecosistemas.
«Editores de base de citosina basados en CRISPR-Cas9 eficientes para bacterias fitopatógenas», se publicó en Communications Biology .
Más información: Chenhao Li et al, Editores de base de citosina basados en CRISPR-Cas9 eficientes para bacterias fitopatógenas, Communications Biology (2023). DOI: 10.1038/s42003-023-04451-8
