El melón (Cucumis melo L.) es un cultivo de hortalizas importante que tiene una extensa historia de cultivo y se ha clasificado en dos subespecies, C. melo ssp. agrestis y C. melo ssp. melo.
por la Universidad Agrícola de NanJing
Estudios anteriores sugieren que las dos subespecies fueron domesticadas de forma independiente, lo que puede haber generado diferentes mecanismos genéticos para el mismo rasgo entre las dos subespecies. Además, la diferencia en su distribución geográfica dio lugar a diversas características entre las dos subespecies, dando forma a la impronta genómica en sus genomas.
El germoplasma silvestre es un recurso genético importante en el mejoramiento de cultivos debido a su alta diversidad genética y resistencia contra enfermedades. Sin embargo, todos los genomas reportados anteriormente se ensamblaron en base al melón cultivado ; el genoma de los tipos de melón silvestre y semisilvestre aún no está disponible. Por lo tanto, ensamblar el genoma del melón silvestre/semisilvestre de alta calidad brindará una oportunidad sin precedentes para el descubrimiento de genes y el mejoramiento de la resistencia en el melón.
Un estudio titulado » El genoma de referencia T2T resuelto con haplotipos destaca la variación estructural subyacente a los rasgos agronómicos del melón » ha sido publicado en Horticulture Research . Yongyang Xu, del equipo de mejoramiento genético de melón del Instituto de Investigación de Frutas de Zhengzhou de la Academia China de Ciencias Agrícolas, cooperó con el profesor Tao Lin de la Universidad Agrícola de China.
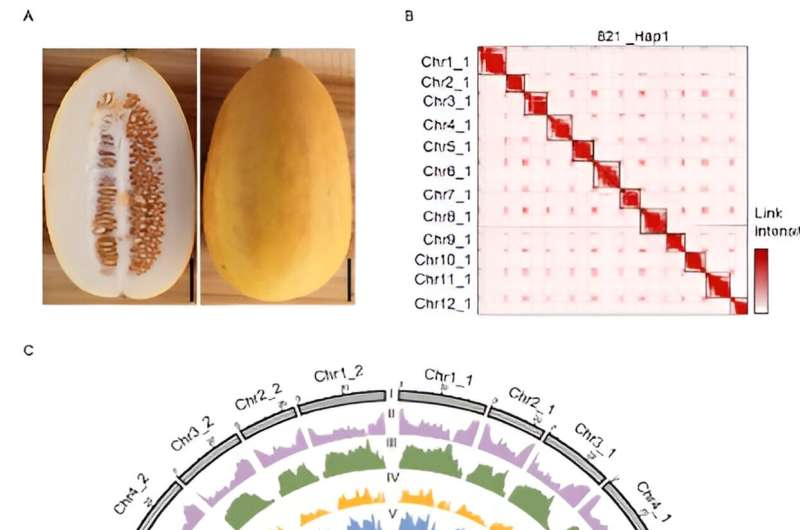
PI 313970 es una accesión derivada de C. melo ssp. agrestis var. acidulus originaria de la India, que poseen alta resistencia al mildiú polvoriento y a una variedad de enfermedades virales (ToLCNDV, CYSDV, CABYV, WmCSV y CuLCrV). La accesión ‘821’ fue autofecundada a partir de PI 313970 durante varias generaciones. Aquí informamos un ensamblaje del genoma T2T a nivel de cromosoma para 821 (C. melo ssp. agrestis var. acidulus), un melón semisalvaje con dos haplotipos de ~373 Mb y ~364 Mb, respectivamente.
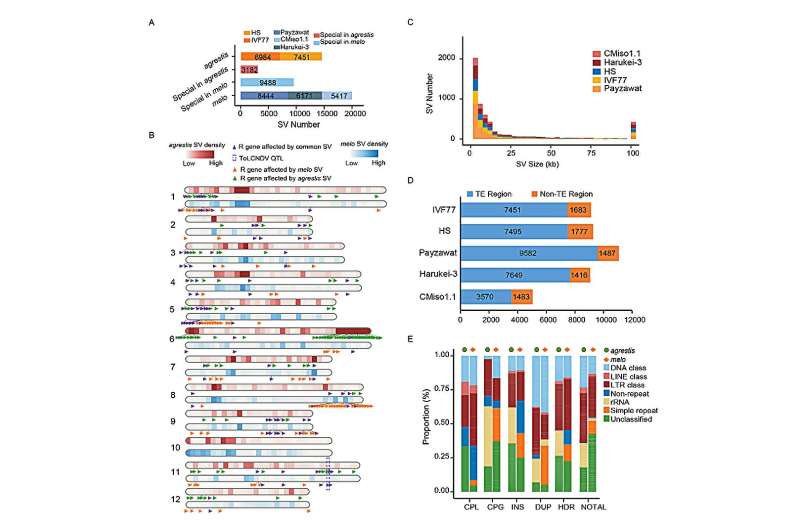
El análisis comparativo del genoma descubrió una cantidad significativa de variantes estructurales (SV) entre los genomas de melo y agrestis, incluida una variación del número de copias ubicada en el locus de resistencia a ToLCNDV en el cromosoma 11, del cual se identificó un gen candidato para la resistencia a ToLCNDV.
El melón es una especie modelo única para estudiar la maduración de los frutos por presentar tipos climatéricos y no climatéricos. Los estudios de asociación de todo el genoma detectaron una señal significativa asociada con la maduración climatérica e identificaron un gen candidato CM_ac12g14720.1 (CmABA2), que codifica una deshidrogenasa/reductasa citoplasmática de cadena corta, que controla la biosíntesis del ácido abscísico. Este estudio proporciona valiosos recursos genéticos para futuras investigaciones sobre el mejoramiento genético resistente en melón.
Más información: Guoli Li et al, El genoma de referencia T2T resuelto por haplotipo resalta la variación estructural subyacente a los rasgos agronómicos del melón, Horticulture Research (2023). DOI: 10.1093/hora/uhad182
