Mentha suaveolens, comúnmente conocida como menta piña, es valorada por su distintivo aroma y propiedades medicinales, que se atribuyen a sus aceites esenciales.
por TranSpread
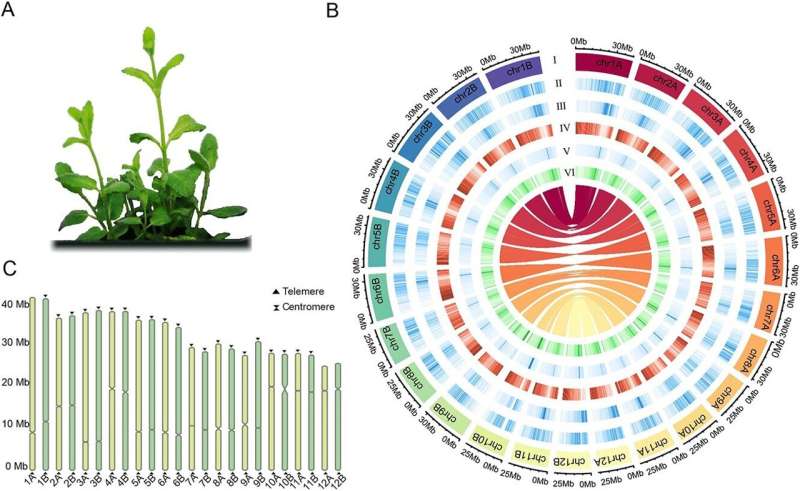
A pesar de su importancia, comprender la base genética de estos rasgos ha sido un desafío debido a la complejidad del genoma de Mentha, caracterizado por una alta heterocigosidad y numerosas variaciones estructurales. Un estudio exhaustivo del genoma de Mentha fue esencial para descubrir los factores genéticos que influyen en sus características únicas.
Investigadores de la Universidad de Medicina Tradicional China de Chengdu, en colaboración con varias otras instituciones, ensamblaron con éxito un genoma libre de espacios y resuelto con haplotipos de Mentha suaveolens, también conocida como menta de piña.
Utilizando tecnologías de secuenciación avanzadas, el equipo arrojó luz sobre la diversidad genética y las variaciones estructurales dentro del genoma, proporcionando un recurso valioso para futuras investigaciones genéticas y de mejoramiento. El estudio se publica en la revista Horticulture Research .
El estudio presenta el primer ensamblaje del genoma resuelto por haplotipos de alta calidad para Mentha suaveolens, con un tamaño de genoma de 414,3 Mb y 31.251 genes codificantes. Al integrar datos de varias plataformas de secuenciación, los investigadores resolvieron dos conjuntos haplotípicos completos, cada uno de ellos casi completamente anotado para telómeros y centrómeros.
En particular, el análisis reveló 41.135 variaciones estructurales, incluidas eliminaciones, inserciones, duplicaciones y translocaciones, muchas de las cuales afectan a los genes implicados en la biosíntesis de terpenoides.
Un hallazgo significativo es el predominio del óxido de piperitenona entre los compuestos volátiles producidos por M. suaveolens, a diferencia del mentol, que es más común en otras especies de Mentha.
El estudio identificó tres genes que codifican la isopiperitenona reductasa (ISPR), una enzima clave en la biosíntesis del mentol, pero encontró que sus bajos niveles de transcripción probablemente conduzcan a la acumulación de óxido de piperitenona.
El Dr. Chi Song, uno de los principales investigadores, afirmó: «La finalización del genoma sin espacios de Mentha suaveolens representa un hito importante en la genómica de las plantas. Este mapa genético integral proporciona una base para explorar los mecanismos moleculares que subyacen a las propiedades únicas de menta piña, que podría conducir a aplicaciones innovadoras en medicina y agricultura».
El ensamblaje del genoma sin espacios de Mentha suaveolens allana el camino para la investigación genética y el mejoramiento para mejorar sus cualidades medicinales y aromáticas. Comprender la base genética de la biosíntesis de terpenoides permite estrategias específicas para optimizar compuestos valiosos como el óxido de piperitenona.
Esta investigación avanza en la genómica de las plantas y tiene un potencial significativo para mejorar el cultivo y el valor comercial de las especies de Mentha.
Más información: Hanting Yang et al, Un ensamblaje del genoma libre de huecos resuelto por haplotipos proporciona información novedosa sobre la diversificación de monoterpenoides en Mentha suaveolens ‘Variegata’, Horticulture Research (2024). DOI: 10.1093/hora/uhae022
