
Las plantas con flores se rigen por el concepto de «cuantos más, mejor» con respecto a sus genomas. En su estado base, son diploides con dos copias del genoma, una de cada padre.
por Massie S. Ballon, DOE / Joint Genome Institute
Tener tres o más copias del genoma de padres adicionales o duplicación, también conocida como «poliploidía», es común entre las plantas con flores; al menos una vez durante su evolución, los genomas de las plantas con flores se multiplican. Con el tiempo, las plantas pierden muchos genes después de tales eventos, devolviendo sus genomas a un estado diploide mientras retienen múltiples copias de algunos genes.https://c728d821a88adce7ef09c5fee09055ba.safeframe.googlesyndication.com/safeframe/1-0-37/html/container.html
Este proceso de poliploidización está en curso y muchas plantas, incluidos los productos agrícolas básicos y los cultivos de materia prima bioenergética candidatos, son poliploides recientes. Los fitomejoradores han aprovechado la poliploidía para aumentar el tamaño de la fruta y la flor y conferir rasgos de tolerancia al estrés. Dada la importancia de la poliploidía en las plantas, los investigadores han buscado conocer los orígenes, la evolución y el desarrollo de los poliploides vegetales. Un equipo multiinstitucional dirigido por investigadores de la Universidad de Zaragoza de España y el Instituto Conjunto del Genoma (JGI) del Departamento de Energía de EE. UU. (DOE), una instalación de usuarios de la Oficina de Ciencias del DOE ubicada en el Laboratorio Nacional Lawrence Berkeley (Berkeley Lab), se basó en un modelo de sistema de césped para obtener respuestas. Su trabajo apareció recientemente en Nature Communications .
El equipo estudió Brachypodium hybridum, un poliploide resultante de la combinación de los genomas de los diploides B. stacei y B. distachyon, un pariente del cultivo de bioenergía candidato al pasto varilla y una planta insignia de JGI. Las tres gramíneas de Brachypodium tienen genomas pequeños, ciclos de vida cortos y pueden transformarse fácilmente, rasgos que facilitan su trabajo bioinformático y en el laboratorio. «Somos afortunados porque las especies progenitoras de Brachypodium hybridum aún existen (mientras que las especies progenitoras diploides de otras plantas poliploides se han extinguido o se desconocen) ”, señaló Pilar Catalán de la Universidad de Zaragoza, una de las autoras principales del artículo.“ Nos ofrece la posibilidad de explorar genómicamente todo el complejo de especies progenitoras y descendientes y, en consecuencia, la evolución de la poliploidía . «
Evaluación de genomas poliploides en un contexto pangenómico

Para el estudio, en parte habilitado por el Programa de Ciencia Comunitaria de JGI, el equipo generó genomas de referencia para B. hybridum y B. stacei, así como múltiples conjuntos de genomas de menor calidad para los tres pastos. Con estos conjuntos de datos, los investigadores crearon un pangenoma que contiene cuatro genomas de B. hybridum y muchos genomas de B. distachyon. Esto se basa en un pangenoma de B. distachyon elaborado previamente .https://googleads.g.doubleclick.net/pagead/ads?client=ca-pub-0536483524803400&output=html&h=280&slotname=5350699939&adk=2265749427&adf=1873531024&w=750&fwrn=4&fwrnh=100&lmt=1599857381&rafmt=1&psa=1&guci=1.2.0.0.2.2.0.0&format=750×280&url=https%3A%2F%2Fphys.org%2Fnews%2F2020-08-brachypodium-polyploid-genome-evolution.html&flash=0&fwr=0&rpe=1&resp_fmts=3&wgl=1&dt=1599857380530&bpp=31&bdt=785&idt=486&shv=r20200901&cbv=r20190131&ptt=9&saldr=aa&abxe=1&correlator=6460579005410&frm=20&pv=2&ga_vid=1344912359.1594327439&ga_sid=1599857381&ga_hid=682921065&ga_fc=0&iag=0&icsg=9086976&dssz=28&mdo=0&mso=0&u_tz=-240&u_his=2&u_java=0&u_h=900&u_w=1600&u_ah=860&u_aw=1600&u_cd=24&u_nplug=3&u_nmime=4&adx=287&ady=2386&biw=1583&bih=757&scr_x=0&scr_y=0&eid=21066897&oid=3&pvsid=2981947906261310&pem=403&rx=0&eae=0&fc=896&brdim=0%2C0%2C0%2C0%2C1600%2C0%2C1600%2C860%2C1600%2C757&vis=1&rsz=%7C%7CpeEbr%7C&abl=CS&pfx=0&fu=8320&bc=31&ifi=1&uci=a!1&btvi=1&fsb=1&xpc=ZH42eI58I6&p=https%3A//phys.org&dtd=499
Un pangenoma es el conjunto no redundante de genes que se encuentran en una especie, pero en este caso, el equipo incluyó dos especies en el pangenoma. Normalmente, los pangenomas son mucho más grandes que cualquier genoma individual, lo que destaca el hecho de que la información genética en un solo genoma de referencia es insuficiente. «Realmente tenemos que evaluar los genomas poliploides en un contexto pangenómico para no sobrestimar la evolución que ocurrió después de la poliploidización», dijo John Vogel, director del programa Plant Functional Genomics del JGI y autor principal del artículo. «La suposición de que todos los genes en un genoma de referencia diploide estaban presentes en el progenitor diploide real de la línea de referencia poliploide simplemente no es cierta en base a estudios previos de pangenoma. Por lo tanto,
Una especie poliploide, dos subgenomas
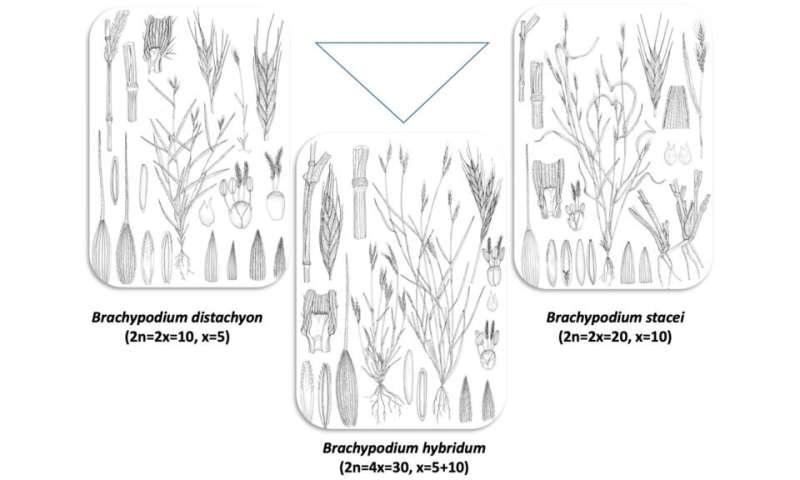
Entre los hallazgos del equipo se encuentra que el poliploide B. hybridum se formó más de una vez, y estos eventos tuvieron lugar con un millón de años de diferencia: el individuo con un padre materno B. distachyon (plastotipo D) hace unos 1,4 millones de años (Mya), mientras que el El plastotipo S fue un evento más reciente (0,14 Mya) con un padre de B. stacei materno. El análisis también reveló que estos dos tipos de B. hybridum no se cruzan. «Los experimentos preliminares de cruces artificiales entre accesiones ancestrales y recientes de B. hybridum produjeron descendientes F1 infértiles (primera descendencia de los padres), lo que plantea la cuestión de si corresponden al mismo resultado de especiación recurrente oa especies diferentes», añadió Catalán.
En general, señaló Vogel, los resultados sugieren que la evolución poliploide está ocurriendo más lentamente de lo esperado. «Aunque es probable que cada caso sea diferente, al menos para B. hybridum está sucediendo mucho más lento de lo que había anticipado con solo una pérdida menor de genes incluso después de un millón de años».
Los genomas de referencia para B. distachyon, B. stacei y B. hybridum están disponibles en el portal de plantas de JGI Phytozome v13 .