Se ha publicado un nuevo artículo titulado «El genoma de referencia completo para la genética y el mejoramiento de la vid (Vitis vinifera L.)» en Horticulture Research.
por la Universidad Agrícola de NanJing
Este genoma PN_T2T sin lagunas (494,87 Mb) ha mejorado significativamente con respecto a las versiones anteriores. La longitud N50 contigua de PN_T2T fue ~ 250 veces mayor que la de 12X.v0 (25,93 Mb frente a 102 kb), y todos los 9429 espacios en 12X.v0 se llenaron en el genoma de PN_T2T. También se corrigieron errores de orientación en 12X.v0 como inversiones y translocaciones con respecto a PN_T2T.
Los autores encontraron que la unidad de repetición de telómeros (TTTAGGG/CCCTAAA) era la más abundante en ambos extremos de cada cromosoma. En cuanto a las regiones centroméricas, los autores encontraron que las repeticiones de 107 pb eran la unidad más abundante en todo el genoma, que tenía 182.620,5 (copias ≥ 2) repeticiones que representaban alrededor del 3,95 % del genoma. Se capturó un total de 343 genes en los centrómeros, y la modificación del ARN, la autofosforilación de proteínas, la integración del ADN, la recombinación del ADN y la fotomorfogénesis aparecieron enriquecidas mientras se exploraban los términos relacionados con el proceso biológico (BP) de estos genes.
Los autores encontraron un total de 377 grupos de genes en el genoma de referencia de la vid; estas duplicaciones a menudo involucran reordenamientos locales y pueden extenderse a megabases con docenas a cientos de genes involucrados. En el cromosoma 16 (23-27 Mb), había 599 genes de dominio enriquecido; en el cromosoma 18 (25~36 Mb), había 1.237 genes enriquecidos en dominios. Entre ellos, muchos dominios fuertemente enriquecidos son parte de los dominios base de resistencia a enfermedades de las plantas (base R), y estos genes R y grupos de genes en las uvas resaltan una gran oportunidad para explorar los mecanismos de defensa de las plantas.
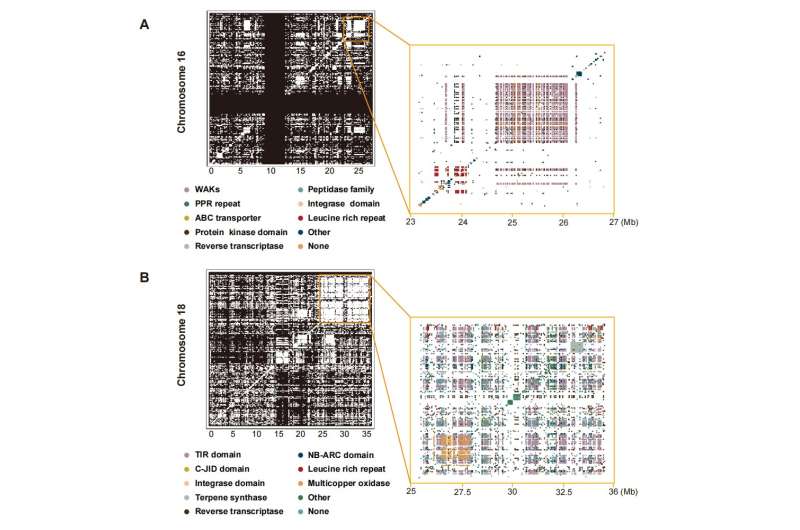
Aunque el genoma de PN40024 es altamente homocigoto (99,8 %), combinado con los datos de resecuenciación de PN40024, los autores encontraron nueve puntos críticos de SNP heterocigotos en los cromosomas. Los resultados mostraron que los términos más significativamente enriquecidos fueron respuesta a la privación de agua, fosforilación de proteínas, división celular , respuesta al estrés oxidativo y respuesta al estrés salino, que estaban estrechamente asociados con actividades fisiológicas clave en las plantas.
En general, las versiones anteriores del genoma de referencia de la vid consistían en miles de fragmentos a los que les faltaban centrómeros y telómeros, lo que limitaba la accesibilidad de la herencia de características agronómicas importantes en estas regiones. Sin embargo, este genoma de referencia sin lagunas, el primer genoma de referencia T2T para árboles frutales para el genoma PN40024, proporciona recursos importantes para la genética y el mejoramiento de la vid.
Más información: Xiaoya Shi et al, El genoma de referencia completo para la genética y el mejoramiento de la vid (Vitis vinifera L.), Horticulture Research (2023). DOI: 10.1093/hr/uhad061
