El género Populus, comúnmente conocido como álamos, álamos y álamos, consta de aproximadamente 30 especies de árboles nativos del hemisferio norte.
por la Universidad Agrícola de NanJing
Debido a sus diversos usos en el paisaje, la agricultura, la bioenergía y la industria, las especies de Populus han sido el foco de muchos programas de mejoramiento genético y mejoramiento genético de árboles.
Las biotecnologías modernas, incluidas la genómica y la ingeniería genética (IG), se han considerado herramientas versátiles para acelerar la domesticación de Populus. Además, desde que se publicó la secuencia del genoma de P. trichocarpa en 2006, seguida de avances en la secuenciación de próxima generación (NGS), los investigadores han podido explorar la base genética de características cruciales en Populus.
La herramienta de edición del genoma CRISPR/Cas9 se ha utilizado ampliamente para crear knockouts en el clon híbrido de álamo «717-1B4» (P. tremula x P. alba INRA 717-1B4). El desarrollo de herramientas alternativas relacionadas con CRISPR, como CRISRPa y la edición de bases, ha demostrado un gran potencial para mejorar la aptitud de las plantas. Sin embargo, la aplicación de estas nuevas herramientas CRISPR en Populus sigue siendo muy limitada.
En mayo de 2023, Horticulture Research publicó el artículo de investigación titulado » Activación de genes basados en CRISPR/Cas9 y edición de bases en Populus «.
En primer lugar, los investigadores construyeron con éxito un sistema de evaluación CRISPRa basado en genes informadores de alto rendimiento en protoplastos. Clonaron los promotores de genes similares a OsTRP u OsCCR1 para generar una construcción informadora.
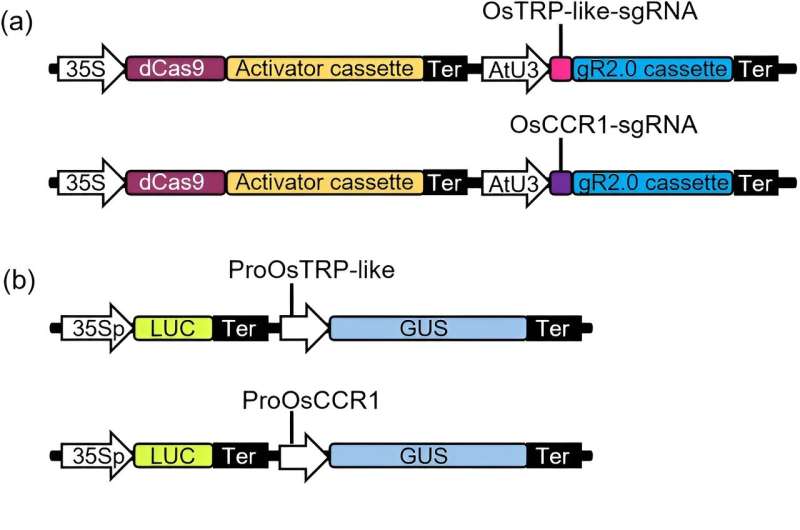
Mediante la cotransfección del vector de activación similar a OsTRP y su vector indicador en protoplastos de Arabidopsis, la proporción de GUS/LUC es seis veces mayor que aquellos sin el vector de activación. También se obtuvieron resultados similares para el gen OsCCR1. Utilizaron este sistema de evaluación para detectar sgRNA en busca de activación genética en protoplastos de Populus.
TPX2 y LecRLK-G son cruciales para el crecimiento y la defensa de las plantas. Clonaron 500 pb de regiones promotoras de TPX2 y LecRLK-G y las insertaron en una construcción informadora. Se seleccionaron seis y tres sgRNA para los genes TPX2 y LecRLK-G, respectivamente. Al cotransfectar las construcciones informadoras y la construcción de activación que contiene cada sgRNA, se evaluó la eficiencia de activación de cada sgRNA mediante las actividades de la enzima GUS. Tanto el sgRNA6 de TPX2 como el sgRNA6 de LecRLK-G se seleccionaron para estudios adicionales de transformación estable.
Luego se establecieron álamos transgénicos de forma estable utilizando los sgRNA identificados. En el clon híbrido de álamo «717-1B4», la expresión del gen TPX2 aumentó entre 1,5 y 2,9 veces. De manera similar, en el clon «WV94», la expresión del gen LecRLK-G aumentó 2 y 7 veces en diferentes eventos.
Estos resultados demuestran que el sistema CRISPR-Act3.0 es capaz de activar genes endógenos TPX2 o LecRLK-G en álamos transgénicos estables. Además, la investigación tuvo como objetivo truncar la proteína PLATZ, vinculada a genes relacionados con enfermedades, mediante la introducción de un codón de terminación prematura utilizando el editor de bases de citosina (CBE) basado en nickasa Cas9 (nCas9) en el clon de álamo «717-1B4».
Después de identificar dos sitios potenciales para introducir codones de parada, los investigadores probaron dos editores de bases (pHEE901(BE3) y A3A/Y130F-BE3) en protoplastos de álamo. Ambos editores indujeron eficazmente mutaciones de C a T en el sistema de protoplastos. Además, pHEE901(BE3) también mostró la edición de bases esperada en álamos transgénicos estables.
En resumen, este estudio presenta una aplicación novedosa de las herramientas CRISPR, específicamente CRISPRa y la edición de bases, para mejorar la función genética en dos especies de Populus. Sorprendentemente, se ha demostrado que el sistema CRISPRa basado en dCas9 activa eficazmente genes en diferentes genotipos de Populus, con posibles implicaciones para cambios fenotípicos en futuras investigaciones.
La investigación establece una base vital para las mejoras genéticas en Populus, mostrando el potencial expansivo de CRISPRa y la edición de bases en especies leñosas, lo que promete avances en el mejoramiento genético de árboles y otras plantas hortícolas.
Más información: Tao Yao et al, Activación genética basada en CRISPR/Cas9 y edición de bases en Populus, Horticulture Research (2023). DOI: 10.1093/hora/uhad085
