El trigo es un alimento básico mundial y desempeña un papel fundamental en los medios de vida de miles de millones de personas.
por Máxima Prensa Académica
Aunque los ARN largos no codificantes (lncRNA) han sido reconocidos como reguladores cruciales de numerosos procesos biológicos, nuestro conocimiento de los lncRNA asociados con el desarrollo del grano de trigo (Triticum aestivum) sigue siendo mínimo.
Seed Biology publicó un artículo en línea titulado » Un atlas completo de ARN largos no codificantes proporciona información sobre el desarrollo del grano en el trigo » el 4 de septiembre de 2023.
Para dilucidar el panorama de los lncRNA en el trigo , el estudio realizó una secuenciación de ARN específica de cadena de todo el genoma (ssRNA-seq) en el endospermo del grano a los 10 y 15 días después de la polinización (DAP) e integró 545 conjuntos de datos de transcriptoma disponibles públicamente de varias etapas de desarrollo y tejidos. El proceso de análisis se adaptó de un estudio anterior con modificaciones y procesó datos de RNA-seq en tres pasos principales: mapeo, ensamblaje y filtrado.
Los resultados identificaron 20.893 lncRNA en trigo. La caracterización de estos lncRNA indicó una longitud de transcripción promedio de 900 pb y eran predominantemente una estructura de exón único (48%), con una superposición significativa con retrotransposones de repetición terminal larga (LTR, 41,40%).
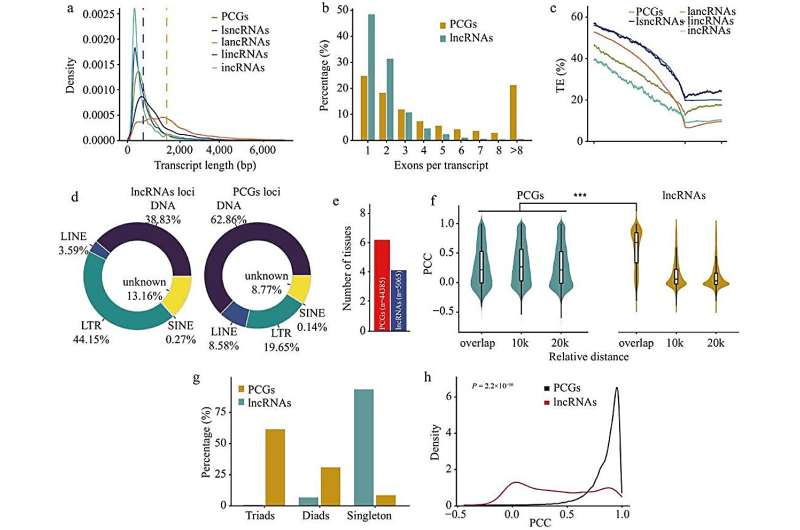
En comparación con los genes codificadores de proteínas (PCG), los lncRNA de trigo exhiben longitudes de transcripción más cortas, menos exones y una mayor expresión específica de tejido que los PCG, y sus patrones de expresión se correlacionaron positivamente con los PCG adyacentes. Además, el análisis de la distribución de los lncRNA en los tres subgenomas del trigo (A, B y D) reveló que el 90,7% de los lncRNA eran exclusivos de un solo subgenoma, lo que sugiere que los lncRNA tienen diferentes trayectorias evolutivas en comparación con los PCG.
Para garantizar un acceso eficiente a estos datos de riqueza, este estudio desarrolló la base de datos integral wLNCdb ( http://wheat.cau.edu.cn/wLNCdb ), que proporciona varias herramientas para explorar perfiles de lncRNA de trigo, incluidos patrones de expresión y redes de coexpresión. , anotaciones funcionales y polimorfismos de un solo nucleótido entre las muestras de trigo.
En particular, utilizando wLNCdb, los autores identificaron el lncRNA TraesLNC1D26001.1, que regula negativamente la germinación de las semillas, ya que su sobreexpresión retrasó la germinación de las semillas de trigo al regular positivamente el 5 insensible al ácido abscísico (TaABI5). Además, este lncRNA parece coexpresarse con genes asociados con la biosíntesis de almidón y proteínas en el trigo, lo que enfatiza su posible papel regulador en el desarrollo del grano y la calidad del uso final.
En resumen, este estudio pionero proporciona un mapa completo de los lncRNA de trigo. El wLNCdb, con su gran cantidad de información y su conjunto de herramientas avanzado, sienta las bases para futuras exploraciones y análisis de las funciones de los lncRNA.
Esta investigación no solo profundiza nuestra comprensión de las funciones de los lncRNA del trigo, especialmente durante el desarrollo de las semillas, sino que también allana el camino para aprovechar este conocimiento para aumentar el rendimiento y la calidad del trigo en el futuro.
Más información: Zhaoheng Zhang et al, Un atlas completo de ARN largos no codificantes proporciona información sobre el desarrollo del grano en el trigo, Seed Biology (2023). DOI: 10.48130/SeedBio-2023-0012
