Un enorme meteoro supuso la perdición para la mayoría de los dinosaurios hace 65 millones de años. Pero no todos. Después del evento de extinción, las aves (técnicamente los propios dinosaurios) florecieron.
Los científicos han pasado siglos tratando de organizar y clasificar unas 10.000 especies de aves en un árbol genealógico claro para comprender cómo los últimos dinosaurios supervivientes llenaron los cielos. La secuenciación barata de ADN debería haber simplificado esto, como lo ha hecho para muchas otras especies.
Pero los pájaros estaban dispuestos a engañarnos.
En un par de nuevos artículos de investigación publicados el 1 de abril, los científicos revelan que otro evento ocurrido hace 65 millones de años los engañó sobre la verdadera historia familiar de las aves. Descubrieron que una sección de un cromosoma pasó millones de años congelada en el tiempo y se negó a mezclarse con el ADN cercano como debería haberlo hecho.
Esta sección, apenas el 2% del genoma de las aves, convenció a los científicos de que la mayoría de las aves podrían agruparse en dos categorías principales, con los flamencos y las palomas como primos evolutivos. El árbol genealógico más preciso, que da cuenta de la sección engañosa del genoma, identifica cuatro grupos principales e identifica a los flamencos y las palomas como parientes más lejanos.
«Mi laboratorio ha estado solucionando este problema de la evolución de las aves durante más tiempo del que quiero pensar», dijo Edward Braun, Ph.D., autor principal del artículo publicado en Proceedings of the National Academy of Sciences y un Profesor de biología en la Universidad de Florida. «No teníamos idea de que habría una gran parte del genoma que se comportaría de manera inusual. Nos topamos con ello».
Braun supervisó a un equipo internacional de colaboradores dirigido por Siavash Mirarab, profesor de ingeniería informática en la Universidad de California en San Diego, para publicar su evidencia de que este trozo pegajoso de ADN enturbió la verdadera historia de la evolución de las aves. Mirarab y Braun también contribuyeron a un artículo complementario publicado en Nature que describe el árbol genealógico de aves actualizado, dirigido por Josefin Stiller de la Universidad de Copenhague.
Ambos artículos forman parte del proyecto de genómica aviar B10K dirigido por Guojie Zhang de la Universidad de Zhejiang, Erich Jarvis de la Universidad Rockefeller y Tom Gilbert de la Universidad de Copenhague.
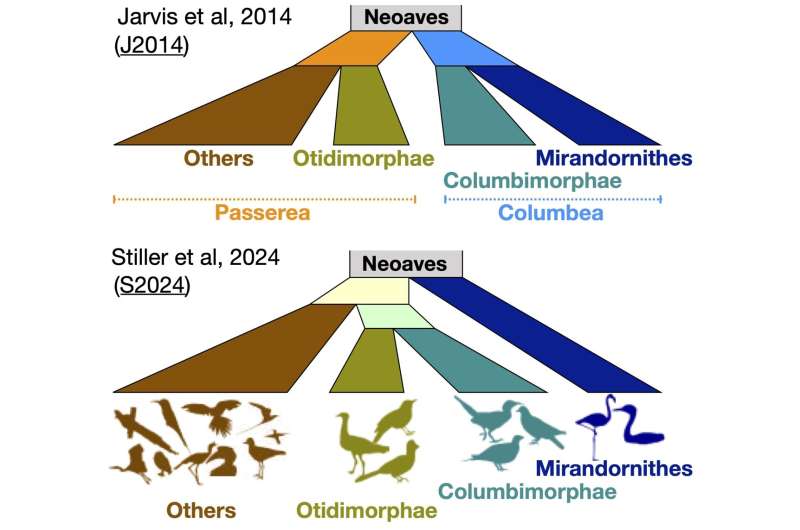
Hace diez años , Braun y sus colaboradores armaron un árbol genealógico de los Neoaves, un grupo que incluye la gran mayoría de especies de aves. Basándose en los genomas de 48 especies, dividieron a los Neoaves en dos grandes categorías: palomas y flamencos en un grupo, todos los demás en el otro. Al repetir un análisis similar este año utilizando 363 especies, surgió un árbol genealógico diferente que dividió a las palomas y los flamencos en dos grupos distintos.
Con dos árboles genealógicos mutuamente excluyentes en la mano, los científicos buscaron explicaciones que les dijeran cuál árbol era el correcto.
«Cuando observamos los genes individuales y el árbol que sustentaban, de repente se dio cuenta de que todos los genes que sustentan el árbol más viejo están todos en un solo lugar. Eso es lo que comenzó todo», dijo Braun.
Al investigar este lugar, el equipo de Braun notó que no estaba tan mezclado como debería haber estado durante millones de años de reproducción sexual. Al igual que los humanos, las aves combinan genes de un padre y una madre en la siguiente generación. Pero tanto las aves como los humanos primero mezclan los genes que heredaron de sus padres al crear espermatozoides y óvulos. Este proceso se llama recombinación y maximiza la diversidad genética de una especie al asegurarse de que no haya dos hermanos iguales.
El equipo de Braun encontró evidencia de que una sección de un cromosoma de ave había suprimido este proceso de recombinación durante unos pocos millones de años, aproximadamente en la época en que desaparecieron los dinosaurios. No está claro si el evento de extinción y las anomalías genómicas están relacionados.
El resultado fue que los flamencos y las palomas se parecían entre sí en este trozo de ADN congelado. Pero teniendo en cuenta el genoma completo, quedó claro que los dos grupos están relacionados más lejanamente.
«Lo sorprendente es que este período de recombinación suprimida podría inducir a error en el análisis», dijo Braun. «Y como podría engañar al análisis, en realidad fue detectable más de 60 millones de años en el futuro. Esa es la parte interesante».
Un misterio así también podría estar acechando en los genomas de otros organismos.
«Descubrimos esta región engañosa en las aves porque dedicamos mucha energía a secuenciar los genomas de las aves «, dijo Braun. «Creo que hay casos como este de otras especies que simplemente no se conocen en este momento».
Más información: Mirarab, Siavash, Una región de recombinación suprimida engaña a la filogenómica neoaviana, Actas de la Academia Nacional de Ciencias (2024). DOI: 10.1073/pnas.2319506121 . doi.org/10.1073/pnas.2319506121
